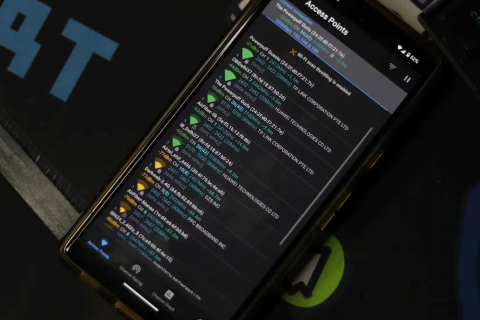
Cómo recuperar el acceso al disco duro, solucionar el error de no poder abrir el disco duro
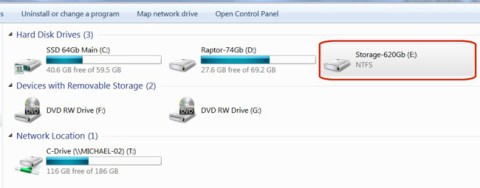
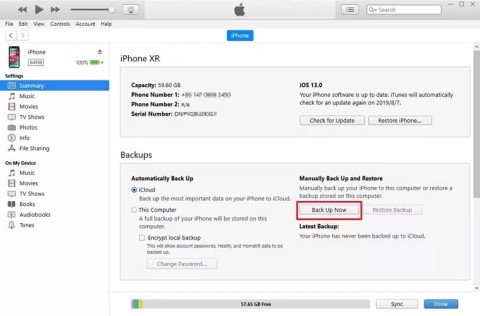
En este artículo, te guiaremos sobre cómo recuperar el acceso a tu disco duro cuando falla. ¡Sigamos!
Un equipo de investigación dirigido por el biólogo sintético Xavier Duportet, cofundador de la empresa de biotecnología Eligo Bioscience, ha diseñado con éxito una herramienta de edición genética que puede modificar la población bacteriana en la microbiota intestinal de ratones vivos.
Los resultados experimentales iniciales mostraron que la herramienta ayudó a modificar el gen objetivo en más del 90% de las colonias de Escherichia coli dentro de los intestinos de ratones vivos. Escherichia coli es un tipo de bacteria que normalmente vive en los intestinos de humanos y animales. Se sabe que la mayoría de las cepas de E. coli causan diarrea leve y transitoria o algunas infecciones intestinales graves que derivan en enfermedades más graves con diarrea, dolor abdominal y fiebre.
Varios estudios previos han utilizado el sistema de edición genética CRISPR—Cas para matar bacterias dañinas en los intestinos de ratones. Pero Duportet y sus colegas querían editar los genes de las bacterias en el microbioma intestinal sin matarlas.
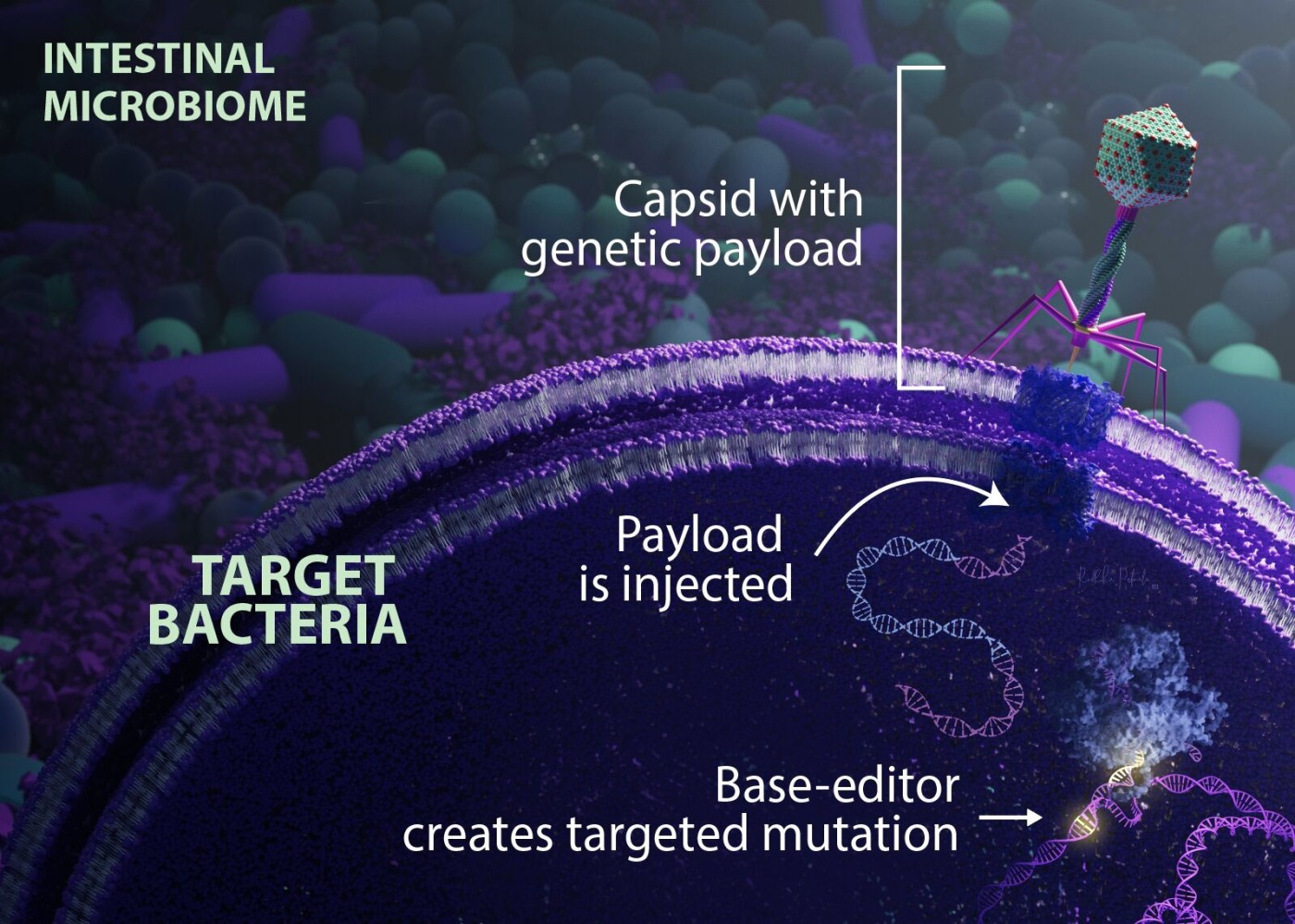
Para lograrlo, los científicos utilizaron un método que consiste en intercambiar una base de nucleótidos por otra (como convertir una A en una G) sin romper la doble cadena de ADN. Hasta la fecha, la mayoría de los métodos existentes no logran modificar lo suficiente la población bacteriana objetivo para ser eficaces. Esto se debe a que los vectores introdujeron únicamente receptores diana que se encuentran comúnmente en bacterias cultivadas en el laboratorio.
Para superar este obstáculo, Duportet y sus colegas diseñaron un vehículo de administración que utiliza componentes de un bacteriófago (un tipo de virus que infecta bacterias) para transportar varios receptores de E. coli expresados en el entorno intestinal. El vector lleva una “herramienta básica de edición genética” que se dirige a genes específicos de E. coli. El equipo también mejoró el sistema para evitar que el material genético suministrado se replique y se propague una vez que está dentro de la bacteria.
El equipo insertó la herramienta básica de edición genética en ratones y la utilizó para cambiar una A por una G en un gen de E. coli que produce β-lactamasa, una enzima que hace que las bacterias sean resistentes a ciertos antibióticos. Aproximadamente ocho horas después de tratar a los animales, aproximadamente el 93% de las bacterias objetivo habían sido editadas genéticamente.
Luego, los investigadores ajustaron la herramienta de edición para que pudiera modificar el gen de E. coli que produce una proteína que se cree que desempeña un papel en algunas enfermedades neurodegenerativas y autoinmunes. El porcentaje de bacterias editadas rondó el 70% dentro de las tres semanas posteriores al tratamiento de los ratones. En el laboratorio, los científicos también pueden utilizar la herramienta para editar cepas de E. coli y Klebsiella pneumoniae que pueden causar infecciones de neumonía. Esto sugiere que el sistema de edición podría adaptarse para abordar diferentes cepas y especies bacterianas.
El logro representa un “gran salto” en el desarrollo de herramientas que pueden modificar las bacterias directamente dentro del intestino, abriendo la posibilidad de combatir enfermedades y al mismo tiempo evitando que el ADN dañino se propague.
El próximo paso para Duportet y sus colegas es desarrollar modelos de ratones con enfermedades inducidas por el microbioma para medir si ediciones genéticas específicas tienen efectos beneficiosos sobre su salud.
En este artículo, te guiaremos sobre cómo recuperar el acceso a tu disco duro cuando falla. ¡Sigamos!
A primera vista, los AirPods parecen iguales a cualquier otro auricular inalámbrico. Pero todo cambió cuando se descubrieron algunas características poco conocidas.
Apple ha presentado iOS 26, una importante actualización con un nuevo diseño de vidrio esmerilado, experiencias más inteligentes y mejoras en aplicaciones conocidas.
Los estudiantes necesitan un tipo específico de portátil para sus estudios. No solo debe ser lo suficientemente potente como para desempeñarse bien en su especialidad, sino también lo suficientemente compacto y ligero como para llevarlo consigo todo el día.
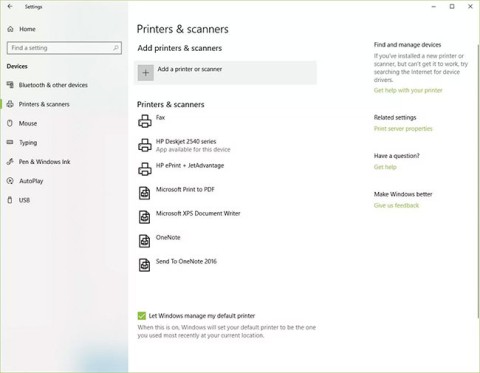
Agregar una impresora a Windows 10 es sencillo, aunque el proceso para dispositivos con cable será diferente al de los dispositivos inalámbricos.
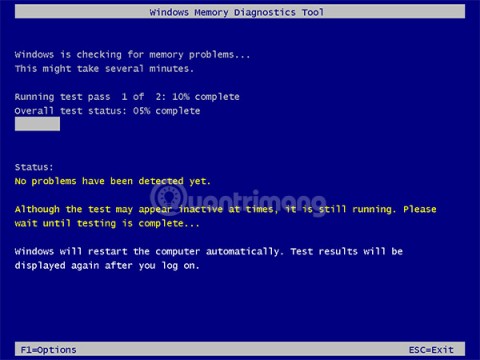
Como sabes, la RAM es un componente de hardware muy importante en una computadora, ya que actúa como memoria para procesar datos y es el factor que determina la velocidad de una laptop o PC. En el siguiente artículo, WebTech360 te presentará algunas maneras de detectar errores de RAM mediante software en Windows.
Los televisores inteligentes realmente han conquistado el mundo. Con tantas funciones excelentes y conectividad a Internet, la tecnología ha cambiado la forma en que vemos televisión.
Los refrigeradores son electrodomésticos familiares en los hogares. Los refrigeradores suelen tener 2 compartimentos, el compartimento frío es espacioso y tiene una luz que se enciende automáticamente cada vez que el usuario lo abre, mientras que el compartimento congelador es estrecho y no tiene luz.
Las redes Wi-Fi se ven afectadas por muchos factores más allá de los enrutadores, el ancho de banda y las interferencias, pero existen algunas formas inteligentes de mejorar su red.
Si quieres volver a iOS 16 estable en tu teléfono, aquí tienes la guía básica para desinstalar iOS 17 y pasar de iOS 17 a 16.
El yogur es un alimento maravilloso. ¿Es bueno comer yogur todos los días? Si comes yogur todos los días, ¿cómo cambiará tu cuerpo? ¡Descubrámoslo juntos!
Este artículo analiza los tipos de arroz más nutritivos y cómo maximizar los beneficios para la salud del arroz que elija.
Establecer un horario de sueño y una rutina para la hora de acostarse, cambiar el despertador y ajustar la dieta son algunas de las medidas que pueden ayudarle a dormir mejor y despertarse a tiempo por la mañana.
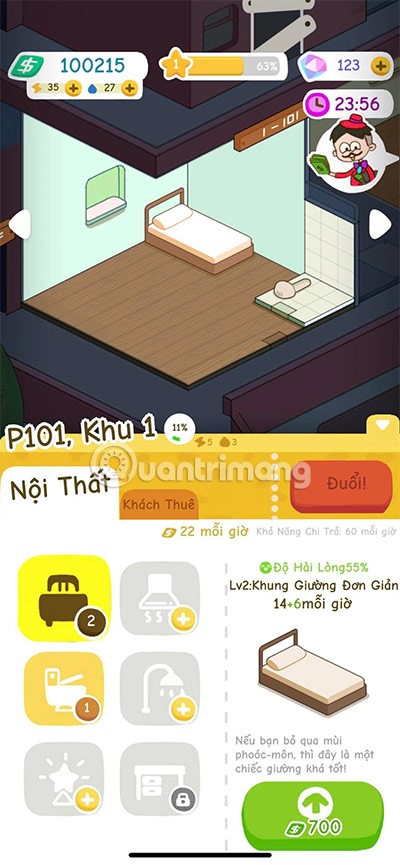
¡Alquiler por favor! Landlord Sim es un juego de simulación para dispositivos móviles iOS y Android. Jugarás como propietario de un complejo de apartamentos y comenzarás a alquilar un apartamento con el objetivo de mejorar el interior de tus apartamentos y prepararlos para los inquilinos.
Obtén el código del juego Bathroom Tower Defense de Roblox y canjéalo por emocionantes recompensas. Te ayudarán a mejorar o desbloquear torres con mayor daño.